Ciencia y Salud, Vol. VI, No. 3, septiembre-diciembre, 2022 • ISSN (impreso): 2613-8816 • ISSN (en línea): 2613-8824 • Sitio web: https://revistas.intec.edu.do/
TEJIDOS FIJADOS EN FORMALINA, EMBEBIDOS EN PARAFINA (FFPE) COMO HERRAMIENTA ÚTIL PARA EXTRACCIÓN DE ÁCIDOS NUCLEICOS DEL SARS-COV-2 EN PRESUNTAS MUERTES CAUSADAS POR COVID-19
Archived Formalin-Fixed Paraffin-Embedded (FFPE) Samples as a Valuable Tool for SARS-CoV-2 Nucleic Acid Extraction in Suspected Deaths Caused by COVID-19
Cómo citar:. Benítez A, Sánchez Marte E, Suero Guzmán C, Vallejo Degaudenzi A, Paulino-Ramírez R. Tejidos fijados en formalina, embebidos en parafina (FFPE) como herramienta útil para extracción de ácidos nucleicos del SARS-CoV-2 en presuntas muertes causadas por COVID-19. cysa [Internet]. 19 de noviembre de 2022 [citado 18 de noviembre de 2022];6(3):35-43. Disponible en: https://revistas.intec.edu.do/index.php/cisa/article/view/2685
Introducción
Un nuevo coronavirus vinculado con el síndrome respiratorio agudo severo asociado a Coronavirus 2 (SARS-CoV-2) se aisló por primera vez a fines de 2019 y, posteriormente, se determinó que era el agente causante de la enfermedad por Coronavirus-19 (COVID-19)1. El número total de muertes a nivel mundial excede los 5 millones de personas desde el inicio de la pandemia2. Solo en las Américas, el SARS-CoV-2 ha provocado más de 2 millones de muertes3.
En la República Dominicana, los primeros casos confirmados de SARS-CoV-2 se notificaron a principios de marzo de 20204, con un aumento fluctuante entre los diferentes puntos críticos, y más de 500 mil casos con una tasa de letalidad que ha rondado entre los 1.25-0.77 %. Los primeros casos notificados se asociaron a personas provenientes de Europa y, posteriormente, fue identificado que la variante circulante predominante en el país era la B.1 que expresaba la mutación D614G.4 El 16 de marzo de 2020 se reportó la primera muerte relacionada, causada por la infección por COVID-19 en República Dominicana.3,5
La infección viral por SARS-CoV-2 se ha asociado a una neumonía altamente infecciosa, con mayor gravedad y tasas de complicación en pacientes con comorbilidades preestablecidas.6,7 Una de las complicaciones asociadas con más frecuencia es un síndrome de dificultad respiratoria aguda grave que se asemeja a los observados en otros coronavirus zoonóticos como las infecciones por SARS‐CoV-1 y MERS‐CoV, pero las nuevas manifestaciones clínicas y características histopatológicas aún se han estado estudiando por sus repercusiones en la terapéutica del síndrome.7-9 El SARS-CoV-2 ha sido clasificado como un organismo del grupo de peligro 3 (HG3) por el Comité Asesor sobre Patógenos Peligrosos (ACDP).10 Las autoridades dominicanas prohibieron la realización de autopsias de rutina para los casos infectados confirmados desde el inicio de la pandemia, lo que explica la limitada disponibilidad de informes con hallazgos patológicos de infección por SARS-CoV-2 en el país11. Por lo tanto, se recogió y almacenó un número muy limitado de tejidos provenientes de autopsias de muertes asociadas a la COVID-19.
En tejidos almacenados se han evaluado diferentes mecanismos para la extracción y preservación de ácidos nucleicos de microbios de interés científico, no obstante, las mismas por su laboriosidad hacen de dicho lento y costoso.12 Debido a la escasa comprensión de los mecanismos asociados con la enfermedad, la detección del SARS-CoV-2 mediante técnicas moleculares a partir de muestras de tejido fijadas con formalina e incluidas en parafina (FFPE) asociadas con el examen microscópico, podría ser una herramienta útil en el proceso de reconocimiento de los mecanismos fisiopatológicos del virus en los tejidos.
El objetivo de este estudio fue detectar el SARS-CoV-2 post mortem mediante la reacción en cadena de la polimerasa con transcripción inversa cuantitativa (qRT-PCR) en muestras de tejidos FFPE y describir la relación con los hallazgos histopatológicos. Asimismo, evaluar el uso de qRT-PCR como técnica post mortem para el diagnóstico de SARS-CoV-2 utilizando FFPE tisular, especialmente en aquellos casos en los que se desconoce la causa de muerte, y los signos y síntomas fueron correspondientes al Síndrome de Distrés respiratorio agudo (SDRA).
Métodos
Examen histopatológico
Este estudio retrospectivo fue efectuado con muestras de tejido recolectadas de autopsias realizadas en el Instituto Nacional de Patología de Santo Domingo. El manejo de cadáveres y el examen de tejidos se completaron de acuerdo con los estándares de bioseguridad rutinarios. Se seleccionaron dos casos que cumplían con los criterios de este estudio: a) historia clínica de dificultad respiratoria grave, b) definición de caso sospechoso de la COVID-19, y c) exposición a viajes asociados con circulación confirmada de SARS-CoV-2 y/o confirmación por detección molecular. El primer caso se diagnosticó como muerte asociada a neumonía adquirida en la comunidad y dificultad respiratoria grave, y el segundo caso se asoció a dificultad respiratoria y antecedentes de viaje asociados con circulación confirmada de SARS-CoV-2. Ambos casos fueron reportados durante el inicio de la pandemia de la COVID-19 en República Dominicana y de los mismos no se obtuvo confirmación molecular definitiva de infección por SARS-CoV-2. Estos dos casos son los únicos reportados como sospechosos a los que se les realizó una autopsia, ya que las regulaciones locales las prohíben en casos confirmados o sospechosos de infección por SARS-CoV-2.
Se seleccionaron un total de 14 áreas respecto al caso 1, y 11 áreas al caso 2. Se tomaron secciones de tejidos del pulmón correspondientes a consolidación macroscópica, zonas de hemorragia y trombo. Además, se obtuvo muestras de corazón, riñón, hígado, páncreas y SNC (Sistema Nervioso Central) y se fijaron en formalina tamponada al 10 %, siguiendo las instrucciones del protocolo. Se cortaron tejidos embebidos en parafina y secciones de 4 µm de espesor y se tiñeron con hematoxilina y eosina (H&E) y tricrómico. Para resaltar los infiltrados inflamatorios mononucleares, se seleccionaron dos secciones y se tiñeron con marcadores inmunohistoquímicos para CD4 + y CD8 + de acuerdo con los protocolos estándar proporcionados por el fabricante biocare medical, llc©. Todas las secciones de tejidos se examinaron con microscopía óptica.
Extracción de material genético
Los ácidos nucleicos se extrajeron de tejidos FFPE archivados durante seis meses, adaptando el método de extracción de columna de centrifugación basado en membrana de sílice. Combinamos dos protocolos, se realizó la lisis usando el Invitrogen PureLink™ Genomic DNA Mini Kit y los pasos restantes de purificación (unión, lavado y elución) usando un protocolo de extracción de ARN menos complejo, el kit radi prepò Swab and Stool DNA / RNA de KHMedical (referencia: MP002) para simplificar la aplicación.
Los lisados de las muestras de FFPE se prepararon cortando hilos de 15 µm y se colocaron aproximadamente 20 miligramos en un tubo de 2 mL. Se añadieron 1 mL de xileno y se agitó vigorosamente durante unos segundos.13, 14 Las muestras se centrifugaron durante tres minutos a 15000 RPM (15596 g) a temperatura ambiente y, con cuidado, se eliminó el sobrenadante evitando la creación de sedimento. Luego se agregó 1 mL de etanol absoluto y se agitó en vórtex para resuspender; posteriormente se centrifugó a 15000 RPM a temperatura ambiente, se retiró el sobrenadante, y se repitió el paso de lavado con etanol. Cada muestra se incubó a 37 °C durante 5-10 minutos para evaporar cualquier residuo de etanol. El resto del proceso de preparación de lisados y la unión, lavado y elución del ARN se realizaron siguiendo el protocolo descrito por ambos fabricantes, a excepción de la adición de la ARNasa proporcionada con el kit Invitrogen PureLink™. Todos los procedimientos se realizaron utilizando una cabina de bioseguridad de clase ii.
Amplificación de ácidos nucleicos
Se obtuvo material de ensayos RT-qPCR de un paso para la amplificación de los genes 2019-CoV E (proteína de la envuelta) y RdRp (ARN polimerasa dependiente de ARN), utilizando el número de catálogo del kit de PCR en tiempo real PowerChek ™ 2019-nCoV: R69900TD. Concretamente, se amplificaron 5 μL de la muestra en una mezcla de reacción de 15 μL que contenía 11 μL de premezcla de RT-PCR y 4 μL de cada mezcla de cebador/sonda, suministrada con el kit. El ensayo se realizó utilizando FAST ABI 7500 Real-Time PCR (Applied Biosystems, Foster City, CA).
Condiciones de ejecución de PCR en tiempo real
Las condiciones para las corridas fueron: un ciclo a 50 °C durante 30 minutos, un ciclo a 95 °C durante 10 minutos, 40 ciclos a 95 °C durante 15 segundos, 40 ciclos a 60 °C durante 1 minuto. Todas las reacciones se realizaron dos veces en experimentos separados.
El canal FAM se utilizó para la detección de fluorescencia de los genes 2019-nCov E y EdRp. Se detectó amplificación del control interno en el canal JOE. Los valores de umbral de ciclo (Ct) ≤ 37 se consideraron positivos. También se incluyó un control negativo correspondiente a los tejidos cardíacos y pulmonares de una autopsia realizada por muerte cardíaca un año antes del inicio de la pandemia.
Resultados
Caso 1
El fallecido corresponde a un varón caucásico de 63 años de ascendencia del norte de Asia ingresado en el servicio de urgencias con dificultad respiratoria repentina. La historia clínica reveló que el comienzo de los síntomas inició cinco días previos al ingreso, con febrículas de 38 °C (100 °C), cuando el servicio de emergencia lo trasladó a un centro de salud el 12 de marzo de 2020. Al ingreso, el diagnóstico presuntivo COVID-19 basado en los antecedentes de viaje y manifestaciones clínicas. El 14 de marzo de 2020, se procedió a tomar un hisopado nasofaríngeo para la detección del SARS-CoV-2. Dicho informe de RT-PCR fue positivo. El manejo clínico de este caso se basó en las guías estandarizadas nacionales del país en ese momento, que incluían hidroxicloroquina, azitromicina y ventilación de apoyo con base en parámetros respiratorios de SPO2 <93 % o PaO2 / FiO2 <300 mmHg. Luego de deteriorarse las condiciones clínicas, el paciente falleció y se realizó una autopsia en el Laboratorio Nacional Forense coordinado por el Ministerio de Salud.
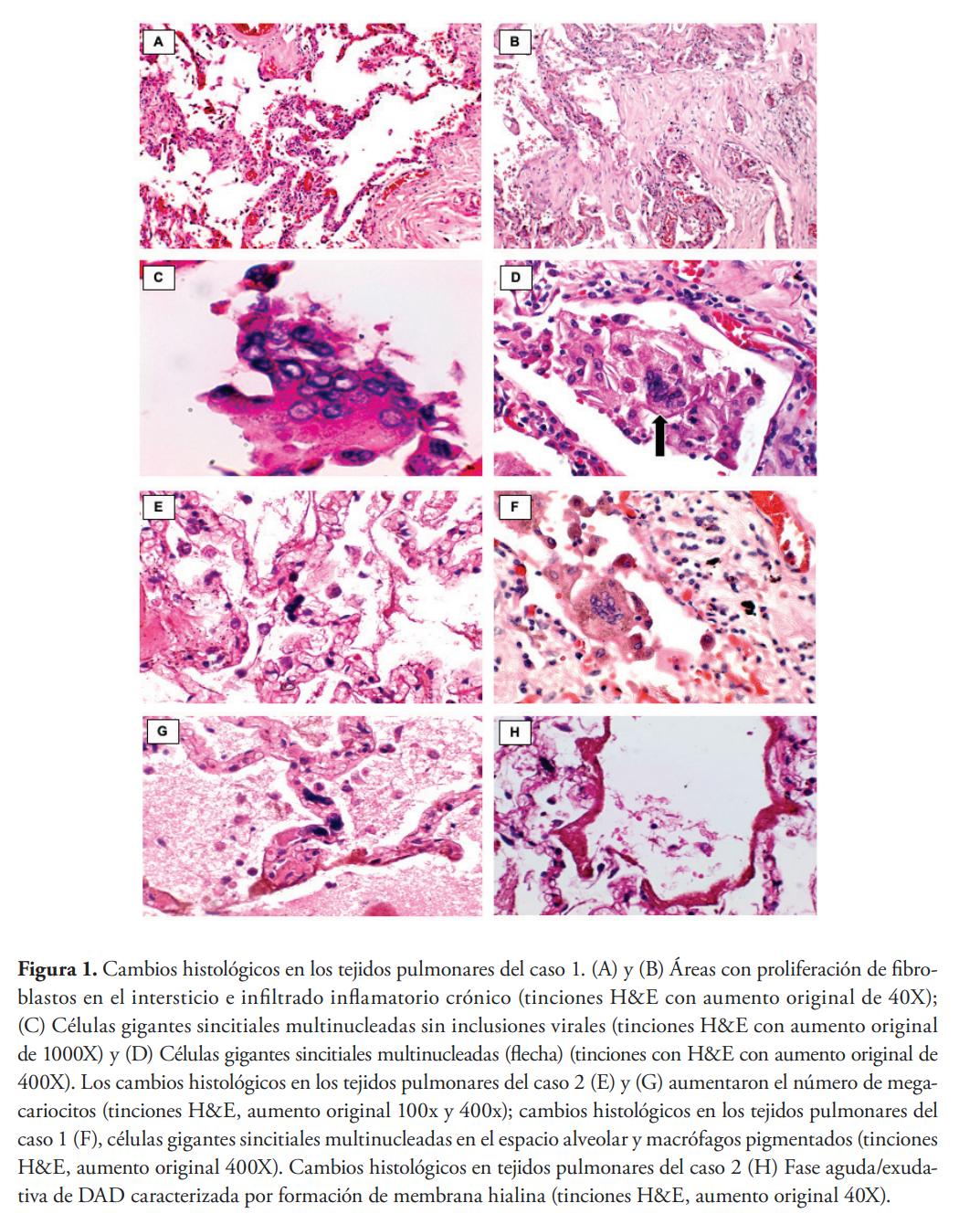
Los hallazgos macroscópicos describieron pulmones pesados, violáceos, con áreas de consolidación en ambas bases pulmonares y antes de la fijación, juntos pesaban 1,270 gramos. El examen microscópico de los pulmones mostró daño alveolar difuso en la fase organizativa (Figuras 1A y 1B) con proliferación de fibroblastos en el intersticio, tabique alveolar congestivo e infiltrado inflamatorio crónico. En otras secciones se encontraron células gigantes sincitiales multinucleadas sin inclusiones virales y macrófagos pigmentados (Figuras 1C y 1D). Además, hubo una proliferación intersticial e intraalveolar de fibroblastos sin infiltración significativa de neutrófilos en los tejidos evaluados en los pulmones. Las zonas hemorrágicas estaban presentes en los tejidos pulmonares centrales y periféricos.
Otros hallazgos significativos se describieron en las muestras de tejidos cardíacos que mostraban miocitos hipertróficos y degenerativos; focales sin un infiltrado inflamatorio linfocítico significativo. Además, el parénquima hepático mostró esteatosis no alcohólica macrovesicular calificada según los estándares de Kleiner como moderada,15 porque menos del 66 % del parénquima estaba afectado. El páncreas pesaba 230 g, con evidencia de pancreatitis aguda hemorrágica con áreas necróticas y de saponificación. Ambos cambios evidenciados por la evaluación microscópica, que muestra necrosis enzimática grasa. Además, se encontró una necrosis tubular aguda en el parénquima renal. En todos los tejidos examinados, el resultado de RT-qPCR fue negativo.
Caso 2
La segunda fallecida era una mujer de 50 años de ascendencia afrocaribeña, con obesidad mórbida. El informe de la autopsia reveló que los pulmones eran pesados y un parénquima firme, la tráquea y las luces bronquio-alveolares estaban completamente llenas por un líquido rosado. El examen microscópico evidenció edema pulmonar con una presencia notable de la formación de la membrana hialina que muestra el Síndrome de Dificultad Respiratoria Aguda (SDRA) en fase temprana con las tinciones H&E respectivamente (Figura 1H). Además, linfocitos moderados, infiltrados en una pequeña cantidad localizados en el intersticio; ocasionalmente se visualizan en el espacio respiratorio y neumocitos denudados. La tinción para marcadores CD4 + y CD8 + mostró que los linfocitos son predominantemente CD4 positivos. Se identificó un aumento en el número de megacariocitos (observado 6/10 HPF Olympus® Cx31) (Figuras 1E y 1H). Además, se observaron escasas células gigantes multinucleadas sin inclusiones virales definitivas en los alvéolos y los conductos alveolares. Se identificaron cambios que sugieren efectos citopáticos virales. En algunas áreas, el intersticio pulmonar se mostró engrosado en comparación con las áreas preservadas.
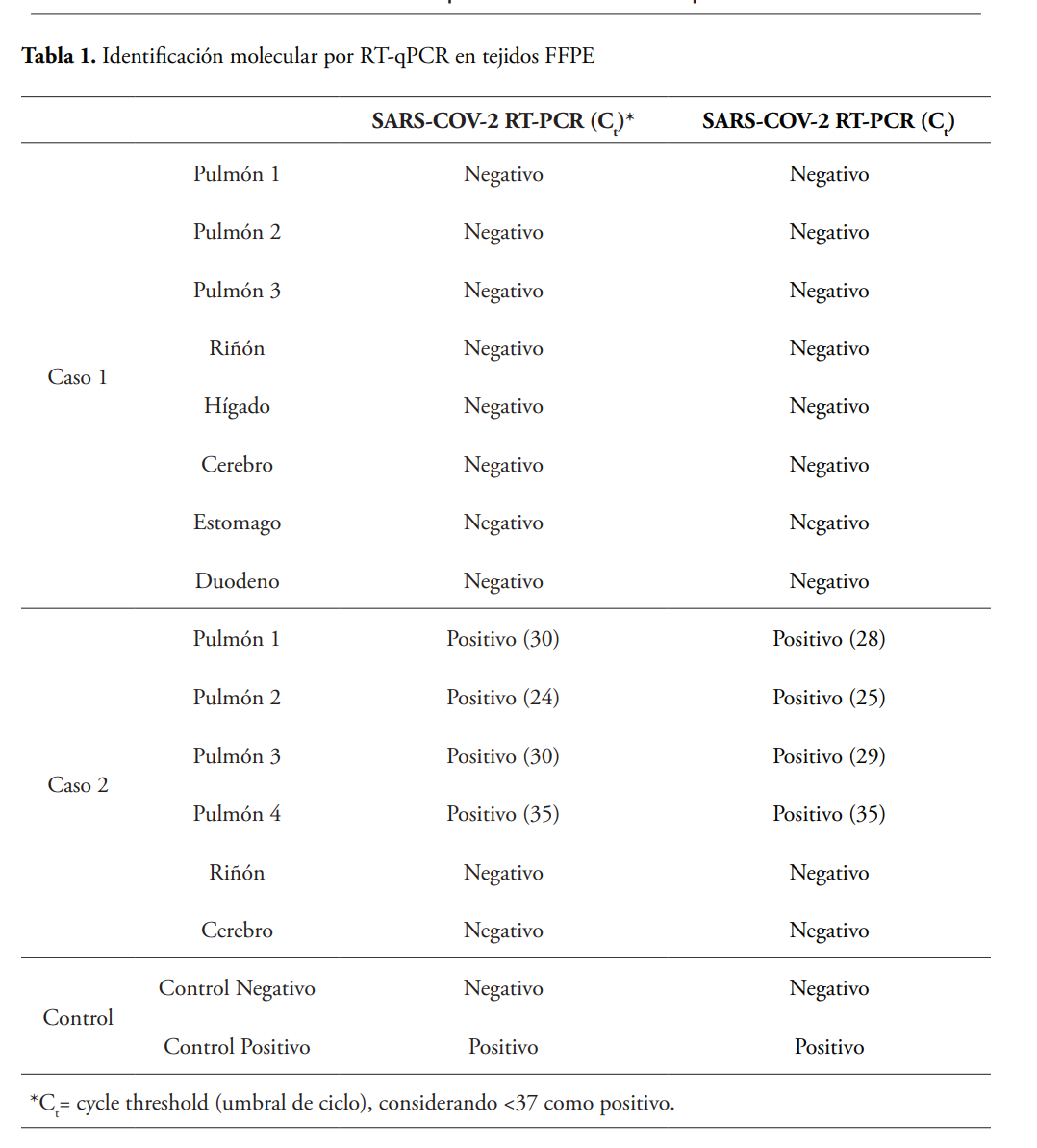
Otros órganos mostraron cambios patológicos inespecíficos de la patología inicial, como necrosis coagulativa del parénquima renal. El parénquima hepático presentaba esteatosis macrovesicular no alcohólica y esta se clasificó como grave porque más del 66 % del tejido se encontraba afectado y en el tejido renal se evidenció necrosis coagulativa por lesión isquémica, probablemente por episodios de hipoperfusión. Todas las muestras de pulmón analizadas fueron positivas para los genes E y RdRp. En otros órganos como hígado, riñón, y cerebro no se identificó la presencia del virus (Tabla 1).
Discusión
En este estudio analizamos los dos únicos casos registrados que se sometieron a autopsia en el país. En estos estuvieron presentes antecedentes de obesidad e hipertensión, similar a las comorbilidades descritas por otros autores.16 Los fenómenos trombóticos y el DAD (Daño alveolar agudo) son características comunes.17 Varios autores han logrado extraer ARN y realizar PCR a partir de tejidos fijados con formalina e incluidos en parafina.18,19
En el caso 1, que corresponde a un estadio clínico más avanzado de SDRA relacionado con infección con características microscópicas, no se identificó material genético viral en ingún órgano. Sin embargo, en las muestras de pulmón correspondientes al caso 2 el material genético viral fue identificado, a pesar de corresponder a un cuadro clínico de menor evolución en el tiempo y con características microscópicas que se relacionan más con un estadio temprano de la enfermedad, aun así, en otros órganos no se identificó. Estos casos sugieren que, a pesar de que las manifestaciones clínicas asociadas al síndrome no fuesen confirmadas por aislados virales, podría coincidir con la evolución del ciclo de vida viral, en el que las manifestaciones deletéreas de la infección pueden perdurar en personas con comorbilidades sin presencia de replicación viral.20 La identificación del virus en tejido pulmonar con TC (tricrómico) en un rango similar, lo confirma tanto en nuestros hallazgos como en los de otros autores.19
De igual forma, estos hallazgos sugieren que es factible realizar una identificación post mortem usando formalina fijada e incluida en parafina para la identificación del SARS-CoV-2, esto indica que la misma podría ser una herramienta útil en casos donde no fue posible tener un diagnóstico concluyente.
Este método puede resultar factible en la práctica de la patología forense y en salud pública, facilitando los ajustes de muerte y calculando la dimensión real de estas asociadas a la COVID-19. Sobre todo, cuando se trata de confirmar o descartar la causa de muerte de aquellos casos que no se estudiaron en el momento oportuno, por ejemplo, al inicio de la pandemia y antes de la notificación del “caso cero”.
Agradecimientos
Los autores quieren agradecer al equipo del Instituto Nacional de Patología en Santo Domingo, por facilitar toda la información sobre el caso y la asistencia en el manejo de las muestras, al Dr. Sergio Sarita Valdez y Santo Jiménez. Todos los autores participaron por igual en la redacción del manuscrito y las conclusiones.
Aspectos éticos
Todos los procedimientos realizados fueron siguiendo los estándares éticos del Comité de investigación institucional y nacional con la declaración de Helsinki de 1964 y sus posteriores modificaciones o estándares éticos comparables. Este estudio fue aprobado por el Comité de Bioética Institucional de la Universidad Iberoamericana cuyo código de aprobación corresponde al número CEI2020-16.
Conflicto de interés
Ninguno de los autores informó conflicto de interés. La asistencia financiera para este estudio fue otorgada a los autores por la Universidad Iberoamericana (UNIBE).
Referencias
- Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, Ren R, Leung KS, Lau EH, Wong JY, Xing X. Early transmission dynamics in Wuhan, China, of novel coronavirus–infected pneumonia. New England Journal of Medicine, 2020;382:1199-207.
- The Center for Systems Science and Engineering (CSSE) at Johns Hopkins University. Coronavirus COVID-19 global. Disponible en https://gisanddata.maps.arcgis.com/apps/opsdashboard/index.html#/bda7594740fd40299423467b48e9ecf6 [Recuperado el 9 de febrero de 2022]
- Dirección General de Epidemiología, Ministry of Health. COVID-19 Special Bulletins. Disponible en https://www.msp.gob.do/web/?page_id=6682 [Recuperado el 9 de febrero de 2022]
- Paulino-Ramírez R, Riego E, Vallejo-Degaudenzi A, Calderon VV, Tapia L, León P, et al. Whole-Genome Sequences of SARS-CoV-2 Isolates from the Dominican Republic. Microbiology Resource Announcements, 2021;10(47):e00952-21.
- Paulino-Ramírez R, Báez AA, Vallejo Degaudenzi A, Tapia L. Seroprevalence of Specific Antibodies against SARS-CoV-2 from Hotspot Communities in the Dominican Republic. Am J Trop Med Hyg. 2020;103(6):2343-6. Doi: 10.4269/ajtmh.20-0907.
- Chan JF, Yuan S, Kok KH, To KK, Chu H, Yang J, Xing F, Liu J, Yip CC, Poon RW, Tsoi HW. A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster. The Lancet, 2020;395(10223):514-23.
- Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, Zhang L, Fan G, Xu J, Gu X, Cheng Z. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. The lancet. 2020;395(10223):497-506.
- Xu Z, Shi L, Wang Y, Zhang J, Huang L, Zhang C, et al. Pathological findings of COVID-19 associated with acute respiratory distress syndrome. The Lancet respiratory medicine, 2020;8(4):420-2.
- Barth RF, Xu X, Buja LM. A call to action: the need for autopsies to determine the full extent of organ involvement associated with COVID-19. Chest, 2020;158(1):43-4.
- Hanley B, Lucas SB, Youd E, Swift B, Osborn M. Autopsy in suspected COVID-19 cases. Journal of clinical pathology, 2020;73(5):239-42.
- Ministerio de Salud Pública de República Dominicana, Organización Panamericana de la Salud. Directrices para el manejo, traslado y disposición final de cadáveres por COVID-19; 2020. Disponible en https://www.msp.gob.do/web/?page_id=6903 [Recuperado el 9 de febrero de 2022].
- Mathieson W, Thomas G. Using FFPE tissue in genomic analyses: advantages, disadvantages and the role of biospecimen science. Current Pathobiology Reports, 2019;7(3):35-40.
- Sekulic M, Harper H, Nezami BG, Shen DL, Sekulic SP, Koeth AT, et al. Molecular Detection of SARS-CoV-2 Infection in FFPE Samples and Histopathologic Findings in Fatal SARS-CoV-2 Cases. Am J of Clin Pathol, 2020;154(2):190-200.
- Martines RB, Ritter JM, Matkovic E, Gary J, Bollweg BC, Bullock H, et al. Pathology and pathogenesis of SARS-CoV-2 associated with fatal coronavirus disease, United States. Emerging infectious diseases, 2020;26(9):2005-15Kleiner DE, Brunt EM, Van Natta M, Behling C, Contos MJ, Cummings OW, Ferrell LD, Liu YC, Torbenson MS, Unalp‐Arida A, Yeh M. Design and validation of a histological scoring system for nonalcoholic fatty liver disease. Hepatology, 2005;41(6):1313-21.
- Djaharuddin I, Munawwarah S, Nurulita A, Ilyas M, Tabri NA, Lihawa N. Comorbidities and mortality in COVID-19 patients. Gaceta Sanitaria, 2021;35:S530-S532.
- Elsoukkary SS, Mostyka M, Dillard A, Berman DR, Ma LX, Chadburn A, et al. Autopsy findings in 32 patients with COVID-19: a single-institution experience. Pathobiology, 2021;88(1):55-67.
- Remmelink M, De Mendonça R, D’Haene N, De Clercq S, Verocq C, Lebrun L, et al. Unspecific post-mortem findings despite multiorgan viral spread in COVID-19 patients. Critical Care, 2020;24(1):1-10.
- Sekulic M, Harper H, Nezami BG, Shen DL, Sekulic SP, Koeth AT, et al. Molecular detection of SARS-CoV-2 infection in FFPE samples and histopathologic findings in fatal SARS-CoV-2 cases. American Journal of Clinical Pathology, 2020;154(2):190-200.
- Wong NA, Saier MH. The SARS-coronavirus infection cycle: a survey of viral membrane proteins, their functional interactions and pathogenesis. International journal of molecular sciences, 2021;22(3):1308.